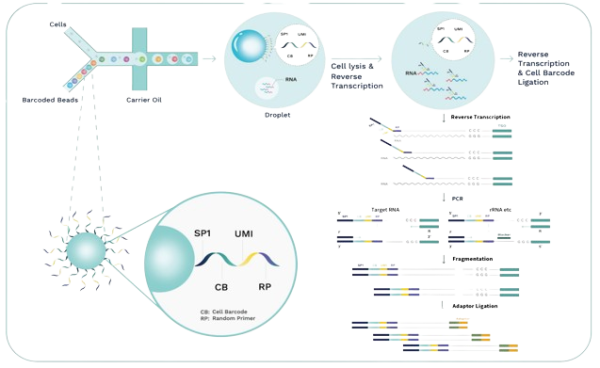
Single Cell Full-length RNA Sequence Transcriptome-seq kit (scFAST-seq)
– Détectez le transcriptome complet sans être limité aux extrémités 3′ ou 5′.
– Obtenez simultanément des informations sur les mutations et l’expression des gènes dans une cellule unique, en établissant la relation entre le génotype et le phénotype.
– Capturez de l’ARN non codant : Outre l’ARNm, l’ARN non codant sans queue polyA 3′ peut également être capturé.
– Franchissez la barrière des espèces : Permet la détection de virus et de procaryotes.
– Améliorez la précision de l’analyse : Permet la détection d’événements de commutation d’isoformes d’ARNm et d’ARNInc.
– Déplétez efficacement l’ARNr : l’inclusion d’un bloqueur d’ARNr permet une déplétion efficace de l’ARNr.
L’utilisation de gouttelettes microfluidiques et de billes à code-barres couplées à des amorces random permet l’obtention d’une distribution et d’une capture aléatoire de l’ensemble du transcriptome en une seule étape. Ce processus ne nécessite qu’une petite quantité d’échantillon. L’inclusion d’un bloqueur d’ARNr permet une déplétion efficace de l’ARNr, et l’obtention d’une plus grande proportion de données utiles.
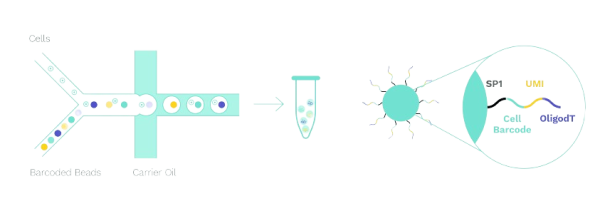
Single Cell 3′ Transcriptome kit
Caractéristiques :
– Compatibilité avec le séquençage Single Cell 3’ du transcriptome (3′ scRNA-seq) et le séquençage Single Cell de l’ARN nucléaire (snRNA-seq). Librairies à double index compatibles avec les séquenceurs Illumina et MGI.
– Générez rapidement 150 000 gouttelettes en 3 minutes avec des taux d’encapsulation élevés notamment pour les échantillons précieux et sensibles.
– Eliminez le gaspillage : Conception unique de la « place chip » pour obstruer les puits vides lors d’un run, afin de les utiliser ultérieurement
Types d’échantillons appropriés :
– Échantillons délicats à faible viabilité cellulaire:tissu cérébral, tissu rétinien, tous les tissus d’individus âgés.
– Tissus nécessitant l’isolation des noyaux : tissus congelés, tissus pancréatiques, neurones, tissus adipeux, etc.
– Etudes de grandes de cohortes où plusieurs échantillons doivent être traités simultanément.
Les cellules uniques et les billes à code-barres sont encapsulées ensemble dans des gouttelettes formant ainsi une émulsion dans le canal en forme de Y. Puis, les molécules d’ARNm libérées par les cellules sont capturées par l’oligo(dT) fixé sur les billes à code-barres.
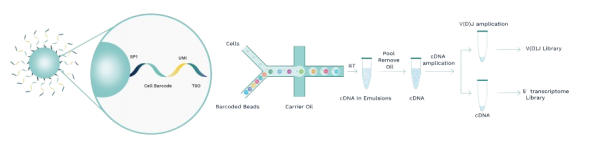
Single Cell Immune Profiling Kit
– Réalisez la couverture complète des séquences V(D)J. Analysez toutes les séquences V(D)J, y compris les séquences CDR3, pour des informations plus complètes.
– Capturez efficacement les séquences V(D)J avec un taux d’alignement supérieur à 90 %. Obtenez des données de capture plus précises et plus fiables.
– Obtenez une représentation fidèle des informations d’appariement des chaînes α/β (légère/lourde) du TCR (BCR)
– Retrouvez les informations d’appariement provenant de la même cellule pour révéler la spécificité récepteur-antigène des cellules immunitaires.
– Profiling simultané du répertoire immunitaire (BCR/TCR) et de l’expression des gènes 5′.
– Réalisez plusieurs librairies à partir d’un seul échantillon, ce qui est plus économique et convient aux échantillons en quantité limitée, tels que les échantillons issus de ponction (biopsie).
Après avoir capturé l’ARNm marqué par des billes à code-barres, deux librairies distinctes (expression génique et répertoire immunitaire) peuvent être construites à partir d’un seul échantillon.